Fortschritt in der RNA-Forschung
18.10.2016Krankheitserregende Bakterien können sich mit Hilfe kleiner RNA-Moleküle gut an ihre Umwelt anpassen. Reguliert wird die Aktivität dieser Moleküle auch durch ein Protein, das Würzburger Infektionsforscher jetzt entdeckt haben.
Für Salmonellen und andere Bakterien, die dem Menschen gefährlich werden können, sind kleine regulatorische RNA-Moleküle lebenswichtig: Dieser RNA-Typus kontrolliert die Aktivität von Genen. Mit ihm können sich die Bakterien schnell an wechselnde Lebens- oder Stressbedingungen anpassen, wie sie typischerweise im Verlauf einer Infektion auftreten – zum Beispiel beim Eindringen in die Blutbahn oder in die Zellen des Menschen.
Bei der Erforschung der kleinen regulatorischen RNA-Moleküle gehört Professor Jörg Vogel, Leiter des Instituts für Molekulare Infektionsbiologie der Universität Würzburg, zu den Pionieren. Mit seinem Team will er Funktionsweise und Wirkungen dieser Moleküle genau verstehen lernen. Seine Arbeiten könnten auch neue Wege zeigen, um Krankheitserreger zu bekämpfen.
ProQ bindet fast 100 regulatorische RNAs
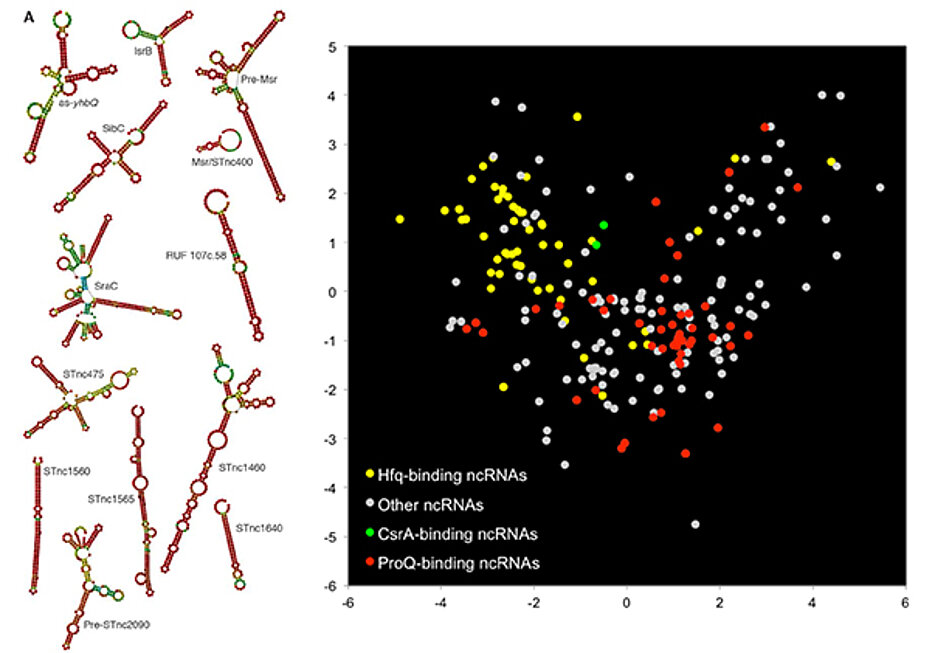
Neue Erkenntnisse aus Vogels Arbeitsgruppe sind jetzt im Magazin PNAS veröffentlicht: Bislang waren zwei Proteine (Hfq und CsrA) bekannt, die sich eng an die regulatorischen RNA-Moleküle der Bakterien binden und deren Aktivitäten beeinflussen. Nun hat das Würzburger Team mit einer von ihm neu entwickelten Methode ein lange vermutetes drittes Protein entdeckt (ProQ), dessen Funktion in der Zelle bisher unbekannt war.
Experimente zeigten, dass das ProQ-Protein an 98 regulatorische RNAs des Darmbakteriums Salmonella enterica bindet. Insgesamt sind aus diesem Bakterium rund 300 solche RNAs bekannt. ProQ scheint sich außerdem auf RNA-Moleküle mit einer eher komplexen Struktur spezialisiert zu haben.
Im „RNA-Universum“ von Bakterien stellen dieses Protein und die daran gebundenen RNA-Moleküle eine bisher weitgehend unerforschte Klasse von Regulatoren der Genaktivität dar. „Hier wird es besonders spannend herauszufinden, wie ProQ die hochstrukturierten RNAs spezifisch unter den Millionen anderer RNA-Moleküle einer Zelle erkennt“, sagt Jörg Vogel.
PNAS stuft Ergebnisse als bedeutsam ein
Das berichtet der Würzburger Professor gemeinsam mit Alexandre Smirnov, Konrad Förstner, Erik Holmqvist und Regina Günster sowie mit Teams aus Greifswald und Köln in PNAS. Das Magazin stuft die neuen Erkenntnisse als so bedeutsam für die Bakterienforschung ein, dass es in seiner Rubrik „Research Highlight“ gesondert darauf hinweist.
Die Aktivitäten des Proteins ProQ konnten mit einer neuen Technik aufgespürt werden, die von der Würzburger Gruppe entwickelt wurde. „Die bislang verfügbaren Methoden hatten beim Nachweis und der generellen Klassifizierung von RNA-Protein-Interaktionen gewisse Grenzen, die wir hier überwunden haben“, so Professor Vogel. Weil sich die neue Methode prinzipiell bei jedem anderen Organismus anwenden lässt, werden von ihr weitere Fortschritte bei der Erforschung der regulatorischen RNA erwartet.
Smirnov A, Förstner KU, Holmqvist E, Otto A, Günster R, Becher D, Reinhardt R, Vogel J: Grad-seq guides the discovery of ProQ as a major small RNA-binding protein. Proc. Natl Acad. Sci. USA (PNAS), 113 (41): 11591–11596, doi: 10.1073/pnas.1609981113
Kontakt
Prof. Dr. Jörg Vogel, Institut für Molekulare Infektionsbiologie der Universität Würzburg, T (0931) 31-82575, joerg.vogel@uni-wuerzburg.de